Snakemake 入门教程(创建一个简单的工作流)
写在前面
既然写了教程就需要具有普适性,能适合大多数人的胃口,我这部分的内容以及示例主要还是参考了官方教程,但是都是我一步一步跑过的流程,所以会更有印象,送给想学 Snkaemake 但是一直没有去学的朋友们,这些内容对于有生信基础的人来讲,上手会很快,因为很多的生信软件都使用过,写起来也就没有那么晦涩,下面开始~
Snakemake 定义
Snakemake 工作流管理系统是一种用于创建可重复和可扩展的数据分析的工具。
工作流是通过一种人类可读的、基于 Python 的语言来描述的。它们可以无缝扩展到服务器、集群、grid和云环境,无需修改工作流。
最后,Snakemake 工作流可能需要对所需软件的准备,这些软件将自动部署到任何执行环境。
安装
Snakemake 可在 PyPi 上以及通过 Bioconda 和源代码获得。可以使用任意方法安装 Snakemake,我们这里仅介绍使用 Conda 安装
通过 Conda/Mamba 安装
这是安装 Snakemake的推荐方式,因为Conda安装比较简单。
首先,必须已经安装了一个基于 Conda 的 Python3 发行版。推荐的选择是Mambaforge,它不仅提供所需的 Python 和 Conda 命令,而且包括 Mamba,它是强烈推荐的Conda包管理器的极其快速和强大的替代品。默认的 conda 求解器有点慢,有时在选择最新的软件包版本时会出现问题。因此,建议在任何情况下都使用Mamba。
如果不安装 Mambaforge ,也可以直接安装 Mamba
1 | conda install -n base -c conda-forge mamba |
单独安装到一个小环境
1 | $ conda activate base |
安装到单独环境中比较好,为了避免与其他软件包冲突,并通过如下方式进行激活
1 | $ conda activate snakemake |
仅安装必备软件的 snakemake 版本
可以安装仅依赖基本必需软件的 minimal 版本 Snakemake
1 | $ conda activate base |
基础示例
首先先激活 Snakemake ,看各自下载的环境,我是单独创建了一个小环境 所以我进行如下操作:
1 | $ conda activate snakemake |
Snakemake 工作流是通过在 Snakefile 中指定命令来定义的。 命令通过指定如何从输入文件集创建输出文件集,将工作流分解为小步骤(例如,单个工具的应用)。Snakemake通过匹配文件名自动确定命令之间的依赖关系。
下面通过创建示例工作流来介绍 Snakemake 语法。 工作流程来自基因组分析领域。 它将测序reads映射到参考基因组,并在映射reads上调用变体。 本教程不需要您知道这是关于什么的。 尽管如此,我们在以下段落中提供了一些背景知识。
测试数据下载
1 | git clone https://bitbucket.org/snakemake/snakemake-tutorial.git |
1. Mapping reads
第一个 Snakemake 命令将给定样本的reads映射到给定的参考基因组。为此,我们将使用工具bwa,特别是 subcommand 。在工作目录中,创建一个使用您选择的编辑器调用的新文件。官方建议使用Atom编辑器,因为它为 Snakemake 提供了开箱即用的语法突出显示。在 Snakefile 中,定义以下命令:bwa mem Snakefile
第一个 Snakemake 命令将给定样本的reads映射到给定的参考基因组。 为此,我们将使用工具 bwa,特别是子命令 bwa mem。 在工作目录中,使用编辑器创建一个名为 Snakefile 的新文件。 官方建议使用 Atom 编辑器,因为它为 Snakemake 提供了开箱即用的语法突出显示。 在 Snakefile 中,定义以下命令:
首先创建一个名称为 Snakefile 的文件,并输入我下面的内容
1 | rule bwa_map: |
Snakemake rule有一个名称(这里是 bwa_map)和许多指令,这里是 input 、 output 和 shell 。
在 input 和 output 指令之后是那些预计将在命令中使用或创建的文件列表。
在最简单的情况下,这些只是 Python 字符串。 shell 指令后跟一个包含要执行的 shell 命令的 Python 字符串。
在 shell 命令字符串中,我们可以通过**大括号表示法(类似于 Python 格式函数)**引用命令的元素。
在这里,我们通过指定 {output} 来引用输出文件,通过指定 {input} 来引用输入文件。由于命令有多个输入文件,Snakemake 将连接它们,用空格分隔。换句话说,Snakemake 会在执行命令之前将 {input} 替换为 data/genome.fa data/samples/A.fastq。
shell 命令使用参考基因组和reads调用 bwa mem,并将输出通过管道传输到 samtools,后者创建包含比对的压缩 BAM 文件。 samtools 的输出被重定向到命令定义的输出文件中,并带有 >。
执行工作流时,Snakemake 会尝试生成给定的目标文件。可以通过命令行指定目标文件。通过执行
1 | # 试运行 |
在包含 Snakefile 的工作目录中, Snakemake 生成目标文件 mapping_reads/A.bam。 由于我们使用了 -n(或 --dry-run)标志,Snakemake 将只显示执行计划而不是实际执行步骤。 -p 标志指示 Snakemake 打印出生成的 shell 命令以供说明。
为了生成目标文件,Snakemake 以自上而下的方式应用 Snakefile 中给出的命令。 应用命令来生成一组输出文件称为作业。 对于作业的每个输入文件,Snakemake 再次(即递归地)确定可应用于生成它的命令。 这产生了作业的有向无环图 (DAG),其中边代表依赖关系。 到目前为止,我们只有一个命令,作业的 DAG 由单个节点组成。 尽管如此,我们可以执行我们的工作流程
1 | $ snakemake --cores 1 mapped_reads/A.bam |
无论何时执行工作流,都需要指定要使用的核心数。 对于本教程,现在将使用单个内核。 稍后介绍并行化是如何工作的。 完成上述命令后,Snakemake 将不会再次尝试创建mapped_reads/A.bam,因为它已经存在于文件系统中。 Snakemake 仅在输入文件之一比输出文件之一新 或 输入文件之一将被另一个作业更新时重新运行作业。
2. Generalizing the read mapping rule
前面的rule仅适用于在文件 data/samples/A.fastq 中读取。 但是,Snakemake 允许使用命名通配符。 只需用通配符 {sample} 替换第二个输入文件和输出文件中的 A,即可批量读取~ 举例:
1 | rule bwa_map: |
当 Snakemake 通过用适当的值替换输出文件中的通配符 {sample} 确定可以应用此命令来生成目标文件时,它将该值传播到输入文件中所有出现的 {sample},从而确定 结果工作的必要输入。
注意,您的文件路径中可以有多个通配符,但是,为了避免与同一命令的其他作业发生冲突,命令的所有输出文件必须包含完全相同的通配符。
1 | $ snakemake -np mapped_reads/B.bam |
Snakemake 将通过将通配符 {sample} 替换为值 B 来确定可以应用命令 bwa_map 来生成目标文件。在试运行的输出中,可以看到通配符值如何传播到输入文件和 shell 命令中的所有文件名。 还可以指定多个目标,例如:
1 | $ snakemake -np mapped_reads/A.bam mapped_reads/B.bam |
一些Bash语法特别方便。例如,可以选择在一次组合多个目标
1 | $ snakemake -np mapped_reads/{A,B}.bam |
在这两种情况下, Snakemake 只创建输出文件 mapping_reads/B.bam。
这是因为之前已经执行过 mapping_reads/A.bam 并且没有比输出文件更新的输入文件。
可以更新输入文件
data/samples/A.fastq的文件修改日期
1 | $ touch data/samples/A.fastq |
并运行 Snakemake 重新运行作业来创建文件 mapping_reads/A.bam
1 | $ snakemake -np mapped_reads/A.bam mapped_reads/B.bam |
3. Sorting read alignments
对于后面的步骤,我们需要对 BAM 文件中的读取对齐进行排序。这可以通过 samtools sort 命令实现。我们在 bwa_map rule下添加以下rule:
1 | rule samtools_sort: |
此命令将从mapped_reads 文件夹中获取输入文件,并将排序后的版本存储在sorted_reads 目录中。
注意,Snakemake 会在作业执行前自动创建丢失的目录。 对于排序,samtools 需要使用标志 -T 指定的前缀。
在这里,我们需要通配符sample的值。 Snakemake 允许通过 wildcards 对象访问 shell 命令中的通配符,该对象具有带有每个通配符值的属性。
wildcards指通配符,学过类 LINUX 系统的,应该都知道什么是通配符。
*代表任意多个字符
?代表任意单个字符
[ ]代表“[”和“]”之间的某一个字符,比如[0-9]可以代表0-9之间的任意一个数字,[a-zA-Z]可以代表a-z和A-Z之间的任意一个字母,字母区分大小写
–代表一个字符
~用户的根目录
1 | $ snakemake -np sorted_reads/B.bam |
看到 Snakemake 首先运行bwa_map,然后运行samtools_sort来创建所需的目标文件:如前所述,依赖项通过匹配文件名自动解析。
4. Indexing read alignments and visualizing the DAG of jobs
接下来,我们需要再次使用 samtools 来索引已排序的读取比对,以便我们可以通过它们映射到的基因组位置快速访问读取。这可以通过以下命令来完成:
1 | rule samtools_index: |
Snakemake 使用Python 格式mini language来格式化 shell 命令。在 shell 命令中使用大括号 (
{}) 来表示其他内容。在这种情况下,必须加倍对我们上面提到的是bash括号扩展依靠时逃避它们,
例如:
ls {{A,B}}.txt
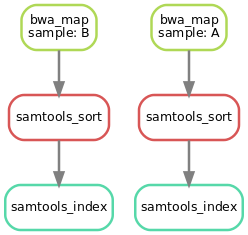
已经完成了三个步骤,现在可以查看生成的有向无环图 (DAG)
1 | $ snakemake --dag sorted_reads/{A,B}.bam.bai | dot -Tsvg > dag.svg |
Snakemake 使用 Graphviz 提供的 dot 命令创建 DAG 的可视化。 对于给定的目标文件,Snakemake 以 dot 语言指定 DAG 并将其通过管道传输到 dot 命令,该命令将定义呈现为 SVG 格式。 渲染的 DAG 通过管道传输到文件 dag.svg 中,如下所示:
5. Calling genomic variants
我们工作流程的下一步将聚合所有样本的映射reads,并共同调用它们的基因组变异。 对于变体调用,我们将结合两个实用程序 samtools 和 bcftools。 Snakemake 提供了一个辅助函数来收集输入文件,帮助我们描述这一步中的聚合。
1 | expand("sorted_reads/{sample}.bam", sample=SAMPLES) |
获取文件列表,其中给定模式sorted_reads/{sample}.bam被格式化为给定样本列表SAMPLES中的值
1 | ["sorted_reads/A.bam", "sorted_reads/B.bam"] |
当模式包含多个通配符时
1 | expand("sorted_reads/{sample}.{replicate}.bam", sample=SAMPLES, replicate=[0, 1]) |
将创建 SAMPLES 的所有元素和列表 [0, 1] 的乘积
1 | ["sorted_reads/A.0.bam", "sorted_reads/A.1.bam", "sorted_reads/B.0.bam", "sorted_reads/B.1.bam"] |
在这里,仅使用expand这个简单的例子。
首先让 Snakemake 知道我们要考虑哪些样本。
Snakemake 从请求的输出反向工作,而不是从可用的输入反向工作。 因此,它不会自动推断所有可能的输出,例如数据文件夹中的 fastq 文件。
Snakefiles 原则上是 Python 代码,通过一些声明性语句来定义工作流。 因此,我们可以在 Snakefile 顶部的纯 Python 中临时定义示例列表:
1 | SAMPLES = ["A", "B"] |
可以将以上命令添加到 Snakefile 中:
1 | rule bcftools_call: |
对于多个输入或输出文件,有时在 shell 命令中分别引用它们会很方便。 这可以通过指定输入或输出文件的名称来完成,例如使用fa=....然后可以在shell命令中通过名称引用这些文件,例如使用{input.fa}。
对于像这样的长 shell 命令,建议将字符串拆分为多个缩进行。 Python 会自动将其合二为一。 此外,您会注意到输入或输出文件列表可以包含任意 Python 语句,只要它返回一个字符串或字符串列表。 在这里,我们调用我们的 expand 函数来聚合所有样本的对齐reads。
6. Using custom scripts
通常,工作流不仅包括调用各种工具,还包含自定义代码,例如计算汇总统计或创建绘图。虽然 Snakemake 还允许您直接在命令中编写 Python 代码。为此,Snakemake 提供了 script 指令。将以下规则添加到您的 Snakefile 中:
1 | rule plot_quals: |
使用此规则,我们最终将生成已分配给文件 calls/all.vcf 中的variant calls的质量分数的直方图。 生成绘图的实际 Python 代码在脚本 scripts/plot-quals.py 中。 在脚本中,命令的所有属性,如 input、output、wildcards 等,都可以作为全局 snakemake 对象的属性使用。 创建文件 scripts/plot-quals.py
1 | import matplotlib |
除了Python脚本之外,还可以使用R脚本;有关详细信息和示例,可以阅读官方教程中的外部脚本部分
7. Adding a target rule
到目前为止,我们总是通过在命令行指定目标文件来执行工作流。除了文件名,如果请求的规则没有通配符,Snakemake还接受规则名作为目标。
因此,可以编写目标规则来收集所需结果或所有结果的特定子集。此外,如果命令行中没有给出目标,Snakemake会将 Snakefile 的第一条规则定义为目标。因此,最好的做法是在工作流的顶部有一个规则all,该rule将所有通常需要的目标文件作为输入文件
1 | rule all: |
把这个rule添加到我们工作流程的顶部。执行 Snakemake 时
1 | $ snakemake -n |
执行此命令将显示用于创建文件的执行计划plots/quals.svg,其中包含并总结了我们所有的结果。
除了 Snakemake 将工作流的第一条规则视为默认目标之外,Snakefile 中的规则顺序是任意的,不会影响作业的 DAG。
总结
生成的工作流程如下所示:
1 | SAMPLES = ["A", "B"] |