HiC-Pro的使用 | HiC辅助基因组组装(一)
定义
软件安装
新版本
建议使用目前最新的3.0.0版本(需要root权限)
安装方法如下:
1 | # 创建conda环境 |
旧版本
如果新版本不适应可以使用旧版本
1 | conda install -c davebx hicpro |
其他安装方法参考官方
软件操作流程
数据准备
-
将Hi-C数据放入对应以样品名命名的目录下。
(注意文件夹名、_R1.fastq.gz._R2.fastq.gz) -
基因组组装结果文件
ln -s PATH=your_assembly.fasta genome.fa
必备文件1-基因组bowtie2索引
1 | bowtie2-build genome.fa genome |
酶切片段文件
1 | /home/lixingze/software/HiC-Pro-3.0.0/bin/utils/digest_genome.py genome.fa -r dpnii -o genome_dpnii.bed |
基因组中序列大小文件
1 | samtools faidx genome.fa |
运行hic-pro
1 | HiC-Pro -c config-hicpro.txt -o analysis -i data |
config-hicpro.txt 配置
其中没有提到的建议使用默认数据操作
1 | ## SYSTEM AND SCHEDULER - Start Editing Here !! |
结果
hic_result/matrix目录
data:存放validpair及其他无效数据文件matrix:存放不同分辨率矩阵文件,
分为raw和iced文件,raw:原始矩阵iced:ice校正后的矩阵后续分析使用,
可以使用HiCPlotter、HiCExplorer出图,或者进行三维基因组学中的部分分析。
pic:存放统计结果图片
stats:存放统计表
hic_result/data目录
allVaildPairs:合并后的valid pairs数据
DEPairs: Dangling end pairs数据
DumpPairs:实际片段长度和理论片段长度不同的数据REPairs:酶切片段重新连接的pairs
FiltPairs:基于min/max insert/fragment size过滤的pairsSCPairs:片段自连的pairs
hic_result/pic目录
plotHiCContactRanges_Example1.pdf有效互作中各类型比例图
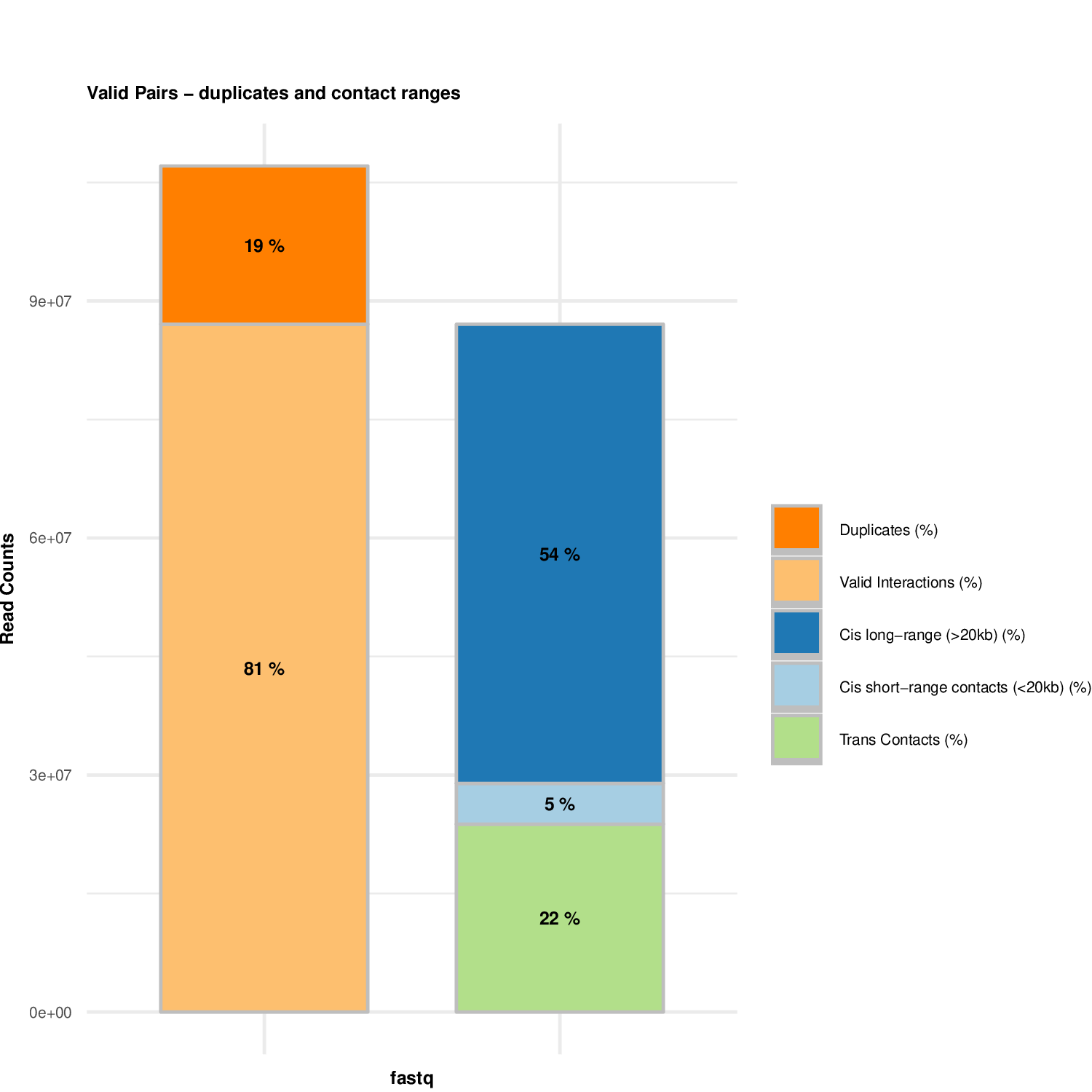
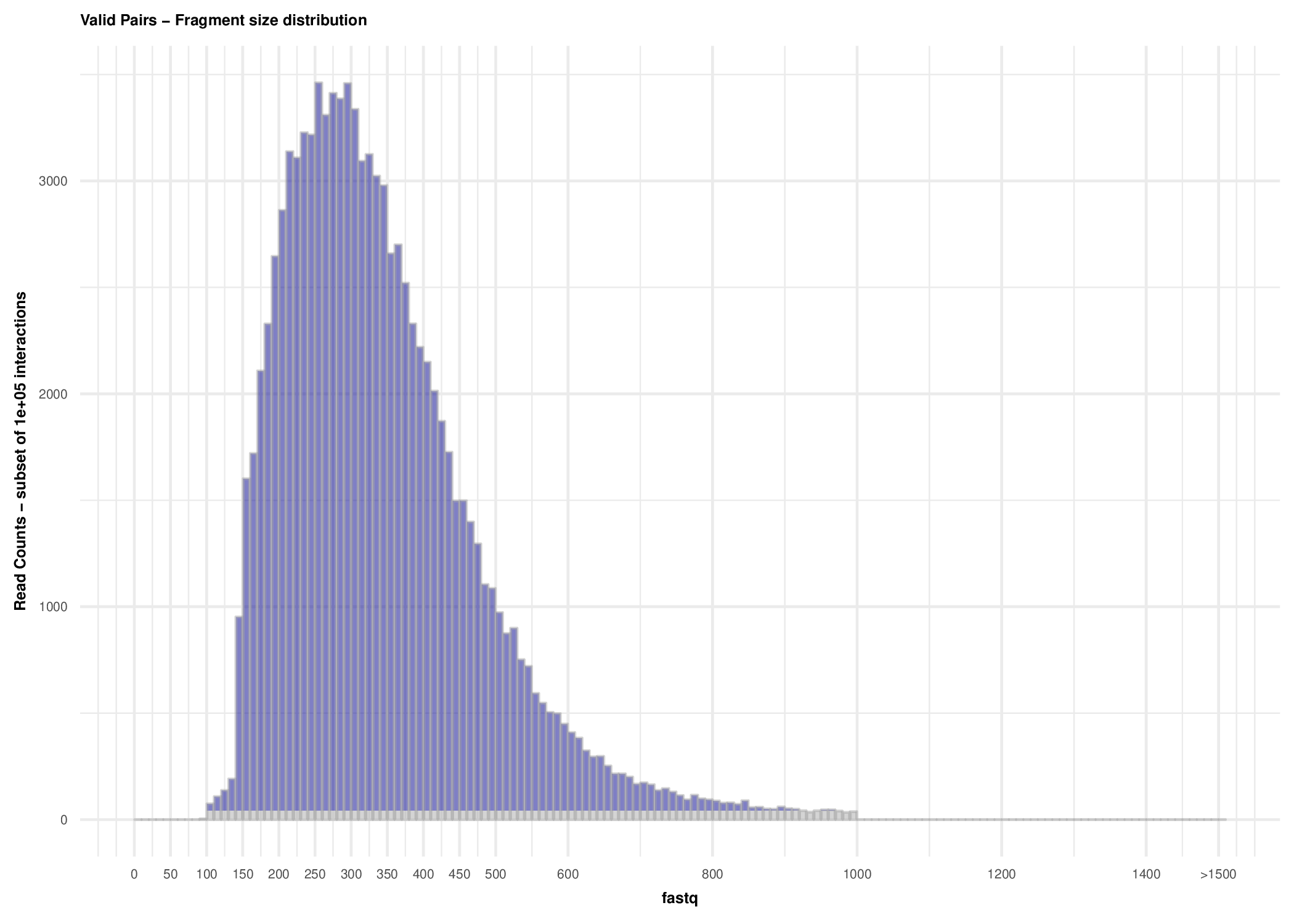
plotHiCFragmentSize_Example1.pdf有效互作的片段大小分布图
plotMappingPairing_Example1.pd合并后双端比对过滤结果图
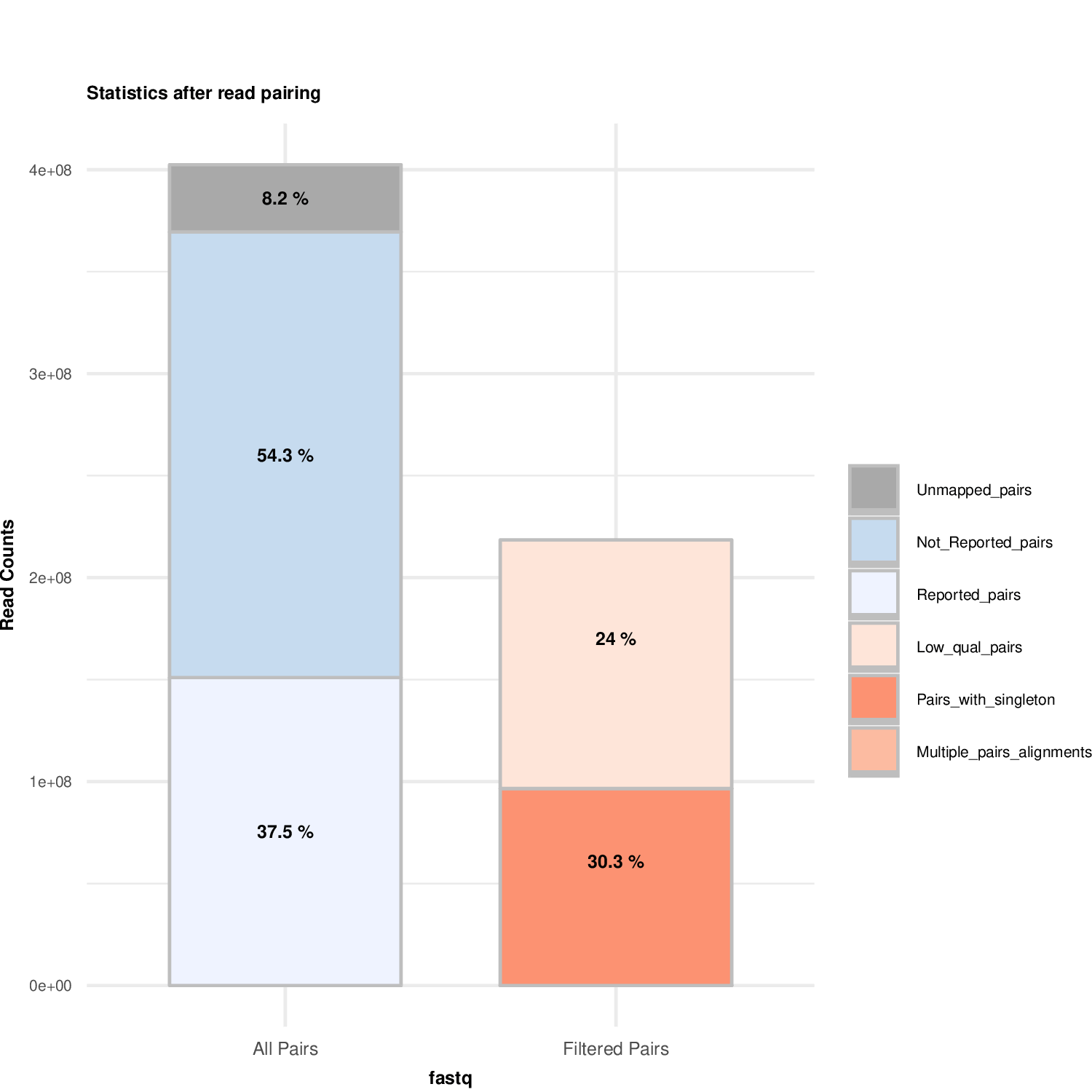 ]
]
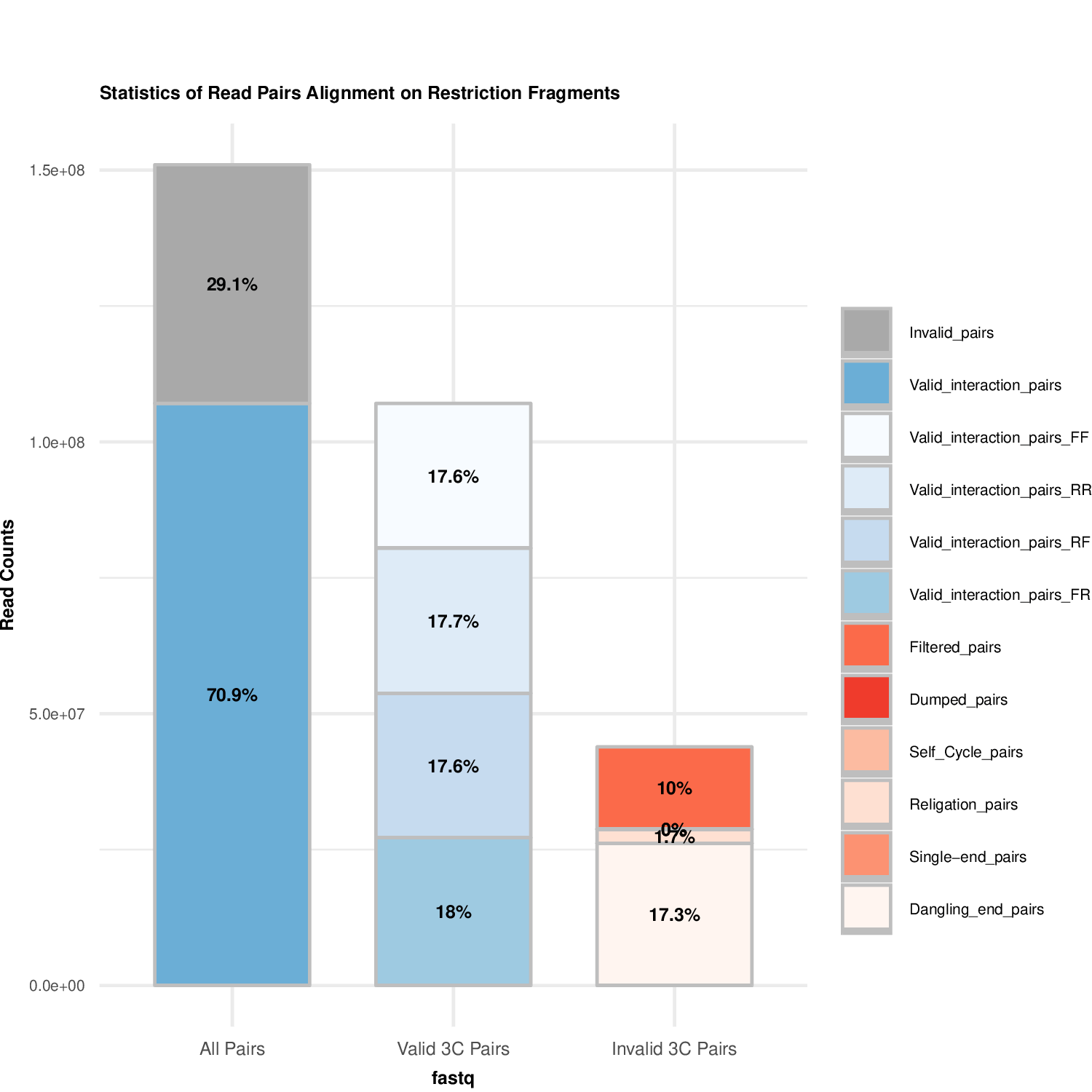
plotHiCFragment_Example1.pdf有效数据过滤结果图
plotMapping_Example1.pdf单端比对过滤结果图
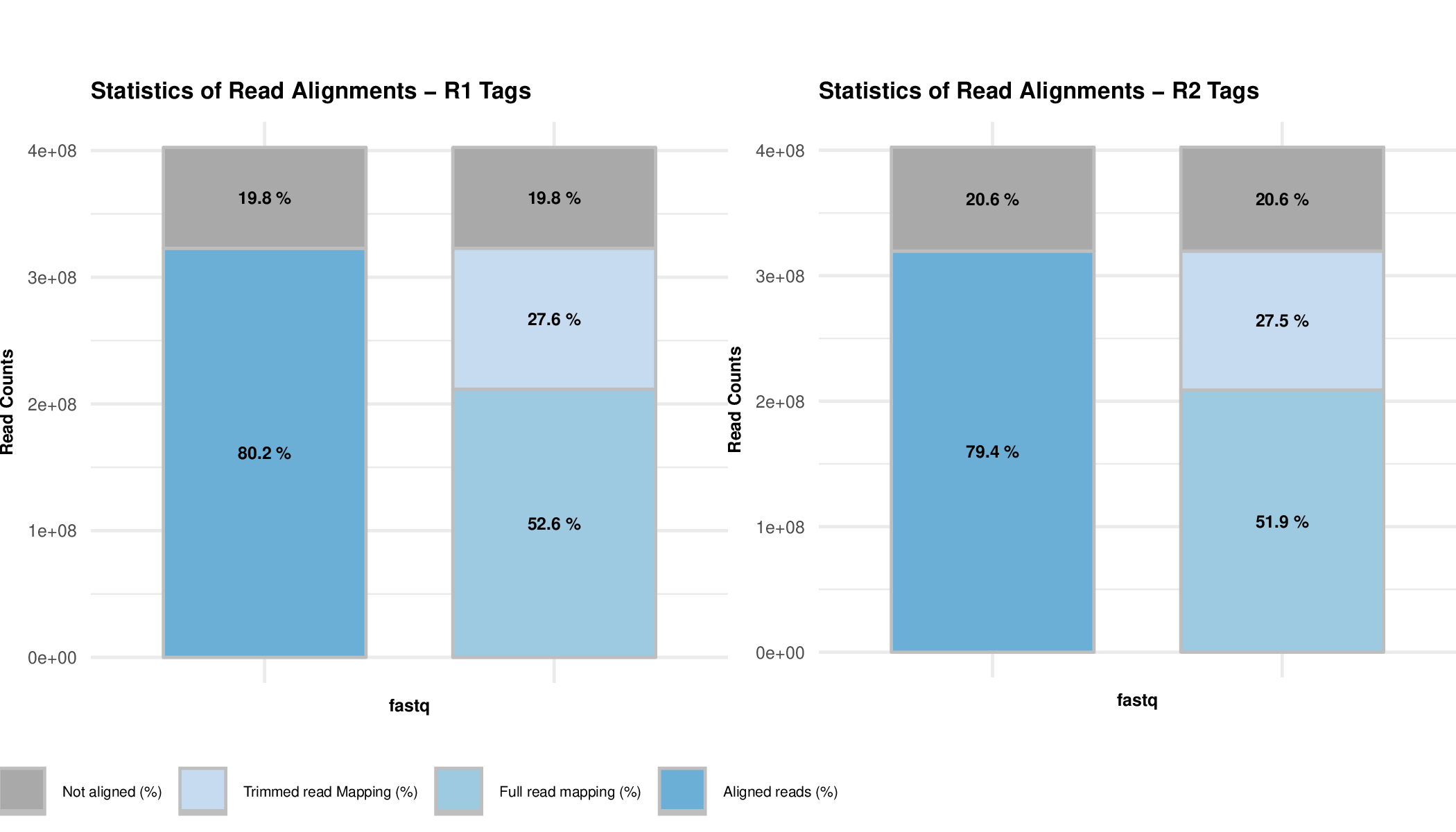
HiC-Pro易报错的地方总结
- 错误一
1 | Exit: Error: Directory Hierarchy of rawdata '/home/lixingze/data/HiC/hicpro/data' is not correct. No '.fastq(.gz)' files detected |
整理reads目录结构
注意:这里在HiCPro的源码中只会读入指定目录的子目录的文件 ,所以将hic测序数据放在子目录下即可
- 错误二
1 | Pairing of R1 and R2 tags ... |
原因可能是之前的bowtie2索引数据有问题造成的,重新跑一次。
- 总结
不同的报错内容很大程度是个人和环境的原因,所以因人而异,不具有普适性













